ИИ успешно предугадывает белковые взаимодействия
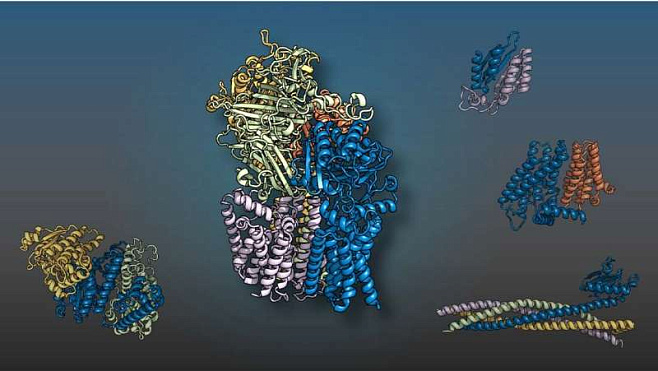
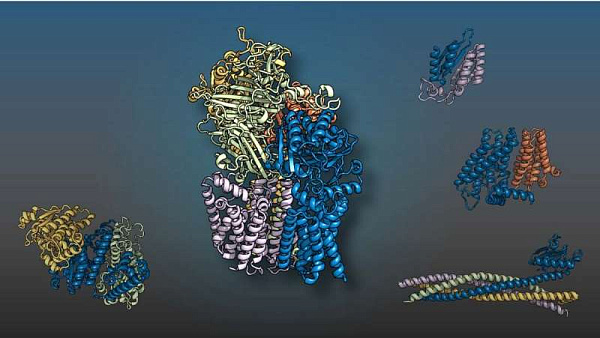
Часто белки выполняют свои задачи в парах или группах, известных как комплексы. Многие такие взаимодействия до сих пор плохо изучены. Создание интерактомов — описаний полного набора взаимодействий молекул в пределах одной клетки — помогло бы лучше разобраться в фундаментальных аспектах биологии, а также пригодилось бы в разработке препаратов, поддерживающих или нарушающих подобные взаимодействия.
В 2020 и 2021 годах компания DeepMind, работающая в развивающемся поле интерактомики, а также учёные из Вашингтонского университета разработали две ИИ-технологии: AlphaFold и RoseTTAFold. Они предугадывают структуры белков по секвенциям производящих их генов. В своей новой работе учёные расширили возможности технологий, смоделировав с их помощью множество белковых комплексов дрожжей.
Всего было выявлено 1505 комплексов, из которых 699 уже были охарактеризованы ранее, 700 были описаны частично, а 106 не были известны. Совместив 3D-модели с информацией о белковых взаимодействиях дрожжей, исследователи получили более точное понимание того, как белковые комплексы связаны с поддержанием и передачей генетической информации, конструкцией и перемещением клеток, метаболизмом, восстановлением ДНК и другими процессами.
Исследователи отметили, что их работа фундаментальна для новой эры структурной биологии, где аналитическое внимание опирается на вычислительные системы.
Фото: UT Southwestern Medical Center
Наука
Антон Тальский



 Российские учёные нашли эффективный способ борьбы с колорадским жуком
Российские учёные нашли эффективный способ борьбы с колорадским жуком Грядущее лунное затмение станет самым долгим за несколько веков
Грядущее лунное затмение станет самым долгим за несколько веков









